Preprocessing includes
– Image analysis and data import
– Background adjustment
– Normalization
– Summarization (for Affymetrix)
– Quality assessment
Practise Data:
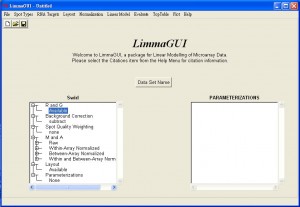
使用limmaGUI匯入two-channel microarray data
參考: http://bioinf.wehi.edu.au/limmaGUI/
A. 下載 Swirl Zebrafish資料
http://bioinf.wehi.edu.au/limmaGUI/Swirl/swirl.zip
B. 安裝 Bioconductor
> source(“http://bioconductor.org/biocLite.R”)
> biocLite()
C. 安裝以下packages: convert, limma, arrayQuality, marray, mclust, hexbin, limmaGUI, sma, tkrplot, R2HTML, statmod
- 程式套件 > 選擇存放處 > CRAN, CRAN(extra), BioC software
- 程式套件 > 安裝程式套件 (或用install.packages(“convert”)
利用affylmGUI匯入two-channel microarray data:
準備動作:
A 安裝estrogen package以取得estrogene dataset:
- 程式套件 > 選擇存放處 > BioC experiment
- 程式套件 > 安裝程式套件
B. 安裝affylmGUI packages:
- 程式套件 > 選擇存放處 > BioC Software
- 程式套件 > 安裝程式套件
Normalization
To identify and remove systematic sources of variation in the measured fluorescence intensities, such as
– different labelling efficiencies of the dyes;
– different scanning parameters;
– sector/print-tip, spatial, or plate effects, etc.
不要做過多的normalization, 因為可能把原本是signal的data弄不見.
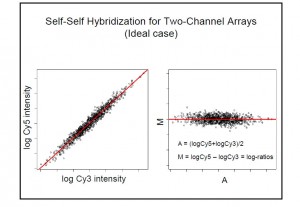
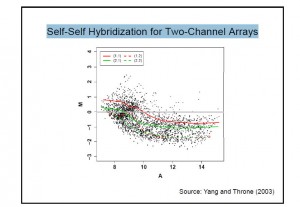
Self-self hybridization: 去探討dye的系統性誤差的典型例子; 使用相同的sample, 不同的染劑, 去做染色就可以知道不同染劑的差異性, 理論上呈現的pattern應該是如下左圖, 但實際上會是長得像右圖的:
所以從以上的例子來看, 矯正(Normalization)的動作變得很重要.
可以用: Within-Array Normalization.
實作:
使用limmaGUI來作Normalization:
Normalization > Select Within-Array Normalization
發佈留言